Indice del volumen Volume index
Comité Editorial Editorial Board
Comité Científico Scientific Committee
BÚSQUEDA DE LA MUTACIÓN Phe-809 EN EL GEN DEL SUSTRATO DEL RECEPTOR DE INSULINA -1 (IRS-1) EN RATAS DIABÉTICAS eSS
Stella Maris Daniele PhD.1, Silvana Marisa Montenegro PhD.2-3,
María Cristina Tarrés PhD.2-3, Stella Maris Martínez PhD 2,3.
1Facultad de Ciencias Bioquímicas, 2Facultad de Ciencias Médicas, 3Consejo de Investigaciones. Universidad Nacional de Rosario.
Rosario, Argentina
smartinez948 @ yahoo.com.mx
Rev Electron Biomed / Electron J Biomed 2010;19-15.
Comentario del revisor Prof. Pilar Muñiz Rodríguez. Titular del Área de Bioquímica y Biología Molecular de la Facultad de Ciencias de la Universidad de Burgos. España PhD.
Comentario del revisor María Jesús Coma MD, PhD. Head of Research Unit. Complejo Asistencial Universitario de Burgos. España
RESUMEN
El gen del sustrato del receptor de insulina IRS-1 juega un rol clave en la transducción de señales de insulina en músculo esquelético. Diversos polimorfismos del gen IRS-1 han sido reportados en estados de insulinorresistencia como obesidad y diabetes tipo 2.
Las ratas eSS desarrollan espontáneamente diabetes tipo 2 magra, con insulinoresistencia caracterizada por hiperglucemia e hiperinsulinemia. Durante el segundo año existe disminución progresiva de la insulinemia y agravamiento del síndrome. Nuestro propósito fue buscar la mutación Phe-809 en el gen IRS-1, que implica sustitución del aminoácido Ser por Phe en el codon 809; dicho sitio se halla conservado en la rata. Esta variante fue identificada en pacientes diabéticos tipo 2. El gen del IRS-1 consta de un solo exón.
Se analizó un fragmento de 281pb del gen IRS-1, que posee 90% de homología con el mismo fragmento en humanos. Se purificó ADN de leucocitos de un pool de sangre de ratas machos eSS y controles Wistar. Se cuantificó y amplificó por PCR utilizando un par de primers para amplificar ese mismo fragmento en el gen IRS-1 en humanos. Se usó sangre humana como control de amplificación. Se empleó el método de Sanger en la secuenciación del fragmento obtenido por PCR, previa extracción del mismo de un gel de agarosa al 2%. Se observó que la rata eSS y las controles mantuvieron la lectura esperada para ese fragmento, no encontrándose la mutación investigada. Se descarta la mutación Phe-809 como origen de la insulinoresistencia de la rata eSS.
PALABRAS CLAVE: mutación Phe-809; gen IRS-1; diabetes tipo 2; ratas eSS
SUMMARY: SEARCH FOR THE PHE-809 MUTATION OF THE INSULIN RECEPTOR SUBSTRATE-1 GENE IN THE DIABETIC ESS RATS
Insulin resistance is a pathophysiological condition involved in the development of type 2 diabetes. The gene encoding Insulin Receptor Substrate - 1 (IRS-1) plays a key role in insulin-mediated signal transduction in both human and animal skeletal muscle. Several polymorphisms of IRS-1 gene have been reported to be associated with states of insulin resistance such as obesity and type 2 diabetes.
The eSS inbred-line of rats spontaneously develops a type 2 diabetes more severe in males than in females. Using eSS males, we investigated the possible presence of the Phe-809 variant in the IRS-1 gene, a site conserved in the rat, which has Ser for Phe at codon 809. This variant has been found in type 2 diabetes patients.
We analyzed a 281pb fragment of the IRS-1 gene, which has 90% homology with the same fragment of IRS-1 in humans. Leukocyte DNA was purified from a pool of blood samples of eSS rats. Wistar rats were used as controls. It was quantified and amplified by PCR with an annealing temperature of 50° C, using a pair of primers employed to amplify the same fragment in the IRS-1 gene in humans. Human blood was used as control of amplification. We employed the method of Sanger sequencing of the fragment obtained by PCR after extraction of an agarose gel 2%. When reading the radiographs, we did not find the mutation. We conclude that eSS rats do not show the Phe-809 mutation in the IRS-1 gene.
KEY WORDS: Phe-809 mutation; IRS-1 gene; type 2 diabetes, eSS rats
INTRODUCCIÓN
La diabetes tipo 2 está relacionada con defectos en la secreción de la insulina y con la resistencia a la acción de dicha hormona, dependiendo su expresión clínica de la interacción de factores genéticos y ambientales1. Tanto en pacientes como en modelos animales, la resistencia a la insulina está íntimamente ligada al desarrollo de diabetes tipo 22-3.
En los últimos años se han identificado en poblaciones humanas mutaciones del gen IRS-1 (sustrato de receptor de insulina) que podrían implicar mayor riesgo genético para diabetes tipo 2 al asociarse con resistencia a la insulina e hiperinsulinemia4.
Se sabe que la activación del receptor de insulina (IR) induce una cascada de reacciones intracelulares que pueden estar alteradas cuando las células expresan receptores de insulina mutados, lo que se traduce en una menor acción de la insulina5-6. IR pertenece a una familia de moléculas receptoras de membrana con actividad tirosina quinasa. El receptor activado fosforila normalmente a una gran familia denominada sustrato del receptor de insulina (IRS), que son mediadores en la acción tisular de la insulina. IRS-1 fue el primer sustrato identificado y representa el prototipo del grupo IRS 7. Es el sustrato mayor para la tirosina quinasa del receptor de insulina y desempeña un rol clave en la transducción de señales de insulina y en el transporte de glucosa mediado por la hormona en las células de los músculos, el hígado y la grasa5,8-9.
La mutación del gen IRS-1 es uno de los factores genéticos que se asocia con predisposición a padecer diabetes tipo 2. En distintas poblaciones del mundo, se han detectado variaciones en el gen de IRS-1 con distinta frecuencia, algunas presentes en pacientes con insulinorresistencia que cursaban con obesidad y diabetes tipo 2 mientras que otras variantes eran silenciosas8,10.
La manipulación genética de ratones en relación con genes candidatos para resistencia a la insulina, ha permitido generar una mutación nula en IRS-1 que induce un importante retardo del crecimiento, resistencia a la insulina, hiperplasia de las células beta e hiperinsulinemia aunque con sólo una ligera intolerancia a la glucosa y sin desarrollo de diabetes manifiesta11. Posteriormente se comprobó que el alelo nulo de IRS-1 induce en los ratones una intensa resistencia a la insulina en las fibras musculares y en el hígado así como hiperplasia marcada de la células beta mientras que ratones con la mutación en el sustrato IRS-2 desarrollan intensa resistencia a la insulina en el hígado, moderada en el músculo y una modesta hiperplasia de las células beta 12.
De los polimorfismos indentificados para IRS-1 en los codones 170, 209, 809 y 97110, cuatro sustituciones de un aminoácido están en sitios conservados tanto en humanos como en ratas y ratones9. La sustitución Ser809 'Phe provocaría alteraciones de su función ya que al estar cercano a Tyr750/751 son sitios de fosforilación. La sustitución Ser809 'Phe fue encontrada exclusivamente en pacientes diabéticos a diferencia de otras cuya prevalencia en pacientes diabéticos no discrepa de la comprobada en controles sanos10.
La rata eSS es una línea con diabetes magra que manifiesta en forma temprana hiperlipidemia e incrementos moderados y progresivos hasta el año de la glucemia y de la insulina plasmática13-14 mientras el páncreas muestra imágenes de nesidioblastosis16. Durante el segundo año de vida, sobre todo en los machos, la insulinemia disminuye con un marcado agravamiento de la hiperglucemia y una dramática disminución en el número y tamaño de los islotes de Langerhans15-16.
El propósito de este trabajo fue investigar la posible existencia de la mutación Phe809 en la rata eSS.
MATERIAL Y METODOS
Se analizó en machos eSS y testigos Wistar (W) un fragmento de 281pb del gen IRS-1 que presenta un 90% de homología con el mismo fragmento del IRS-1 humano.
1. Muestras: Pool de sangre de ratas machos de la línea eSS y de ratas controles W de 12 meses de edad. Se utilizó sangre humana como control de amplificación del fragmento (por PCR) que contiene la mutación buscada.
2. Purificación del ADN genómico:
Se extrajo ADN de leucocitos de sangre obtenida por punción cardíaca por la técnica de Proteinasa K-SDS17-19. Se determinó la concentración de ADN a partir de una alícuota de cada uno de los ADN purificados se las diluyó en un volumen final de 1ml y se realizó un barrido de absorbancias en un espectrofotómetro en la región ultravioleta en un rango de longitudes de onda: 220-300 nm. Teniendo en cuenta que el coeficiente de absorbancia a 258nm para ADN es 50 g/ml, se calculó la concentración de ADN en cada muestra. La relación de absorbancias 258/280nm permitió determinar la pureza de las muestras en cuanto al contenido de proteínas. La integridad del ADN se analizó por electroforesis en geles de agarosa al 1%, en presencia de buffer TBE 1x (pH 8.0), coloreados con bromuro se etidio (10mg/ml de agua) según técnicas convencionales.
3. Amplificación por PCR:
Se amplificó el fragmento de 281pb, utilizando un par de primers (forward y reverse) empleados para amplificar ese fragmento del gen del IRS-1 en humanos.
Secuencia nucleotídica de los primers humanos usados:
- Primer forward: 5 CTCTCCTACTACTCATT
Primer reverse: 5 CAGACCAATAGCCGCCT (En la rata, en el lugar de T hay una C)
En la reacción de PCR se usaron 250 g de ADN genómico, (según concentración); 5 l de buffer 10x, 1 l MgCl2 (concentración final 1,5 mM), 4 l dNTP (concentración final 200 uM), 1 l primers (concentración final 50 pmoles de cada uno) y 0.2 l (0.2 Unidades) GIBCO-BRL de Taq Polimerasa; se completó con H2O hasta un volumen final de 50 l. Se utilizó un termociclador Stuart Sientific teniendo como condiciones de amplificación 95 ºC durante 3 min; 29 ciclos compuestos por 95ºC durante 1 min, 50ºC durante 1 min, por 72ºC durante 1min y otros 72ºC durante 5min.
El tamaño del producto de amplificación de 281pb puede verse en Resultados.
Mediante el método de Sanger se realiza la secuenciación de ADN doble cadena obtenido por amplificación mediante la técnica de PCR. El fragmento de ADN amplificado por PCR se extrajo del gel de agarosa aL 2% después de la corrida electroforética y para ello se usó un kit de extracción rápida del gel ("Concert Gel Extraction Systems" - Gibco BRL).
El método de secuenciación desarrollado por Sanger consiste en la síntesis de ADN in vitro por una ADN polimerasa utilizando como templado cadena simple de ADN. La síntesis se inicia en un solo sitio donde un primer de oligonucleótidos se une al templado específicamente, que actúa como cebador para la reacción de síntesis, ésta se lleva a cabo por incorporación de nucleótidos complementarios, uno de ellos marcado con isótopos radioactivos. La reacción termina por la incorporación de un análogo nucleotídico (dideoxinucleótido, ddNTP) que impide la continuación de la polimerización.
4. Protocolo de marcación del primer:
Se realizó la marcación del primer con ATP32 por medio de la enzima T4 polinucleótido kinasa, del extremo 5 del primer específico al fragmento de ADN doble cadena a secuenciar. Se utilizó como primer de secuencia (10 pmol) 4 l (dilución 1/60); 3 l de ATP32 (10pmol), 1 l 10x Buffer de Kinasa, 0.5 l T4 Polinucleótido Kinasa (5U), llevando a un volumen final con agua de 10 l. Se mezcló e incubó a 37ºC por 30 min y luego se inactivó la kinasa a 90ºC por 3 min. Se centrifugó brevemente y el primer marcado se conservó a -20ºC por el término de una semana.
La mezcla de reacción (Mix) compuesta por 7 l de templado de ADN, 1.5 l de primer marcado con 32P, 5 l de buffer de secuencia 5x, 2.5 l de H2O esteril y 1 l de Taq Polimerasa (5u/ l). Se mezcló, se centrifugó brevemente y se conservó en hielo. Se marcaron 4 tubos de microcentrífuga como A, C, G y T y en cada uno de ellos se colocaron 2 l de mezcla apropiada de d/ddNTP respectivamente (mezcla de terminación A, mezcla de terminación C, mezcla de terminación G y mezcla de terminación T); se agregaron 4 l de Mix a cada tubo. Se colocó una gota de aceite mineral sobre cada mezcla y se centrifugó brevemente a 12000 r.p.m. manteniéndose el tubo en hielo.
5. Programa de ciclado para secuenciación:
Se calentó el termociclador a 95ºC, se colocaron los tubos e inmediatamente se comenzó con el programa de ciclado con 95ºC por 2 minutos, luego, 30 ciclos de 30 segundos (s) a 95ºC (desnaturalización), 30 s a 58ºC (annealing) y 60 s a 70ºC (extensión). Una vez finalizado el ciclado se agregó a cada tubo, 3 l de solución stop que contiene fosfamida y los colorantes que marcan el frente de corrida.
Previamente a la corrida electroforética, las muestras se desnaturalizaron a 90ºC por 5 min y se sembraron 4 l de cada reacción en geles de poliacrilamida al 6% con urea. Finalizada la electroforesis, el gel se fijó con ácido acético al 10% y metanol al 10% por 20 min a temperatura ambiente y se secó por vacío. Las soluciones usadas como buffer de corrida TBE 10x, 54g Tris, 27.5 l ácido bórico, 20ml EDTA (0.5M) y H2O c.s.p. 500ml. La reparación del gel de poliacrilamida al 6% se llevó a cabo con 126g de urea, 17.4g de acrilamida, 0.6g de bisaclilamida, 27ml de TBE 10x y H2O c.s.p. 300 ml.
Para la elaboración de cada gel de secuencia se utilizaron 60ml de mezcla, 60 l de TEMED y 120 l de una solución recientemente preparada de persulfato de amonio (500mg/ml). La electroforesis de 50 x 35cm de Life Technologies SA (1200 - 1500V, 60 Watts, mA libres).
Tras finalizar el recorrido electroforético, se realizó la radiografía y posterior lectura de la secuencia.
RESULTADOS
Amplificación por PCR: la figura 1 muestra el recorrido electroforético en gel de agarosa al 2% del producto de amplificación por PCR del fragmento de 281pb del gen del sustrato del receptor de insulina (IRS-1). En la calle 1 se observa el control negativo, en la calle 2 el fragmento de IRS-1 de las ratas macho eSS, en la calle 3 el fragmento IRS-1 de las ratas Wistar y en la calle 4, el fragmento de IRS-1 humano.
Secuenciación del ADN doble cadena por el método de Sanger: en la posterior secuenciación del producto de amplificación correspondiente al fragmento de 281pb del gen del IRS-1 no se halló la mutación puntual Phe-809 (TCT' TTT) en las ratas eSS ni en las ratas controles Wistar.
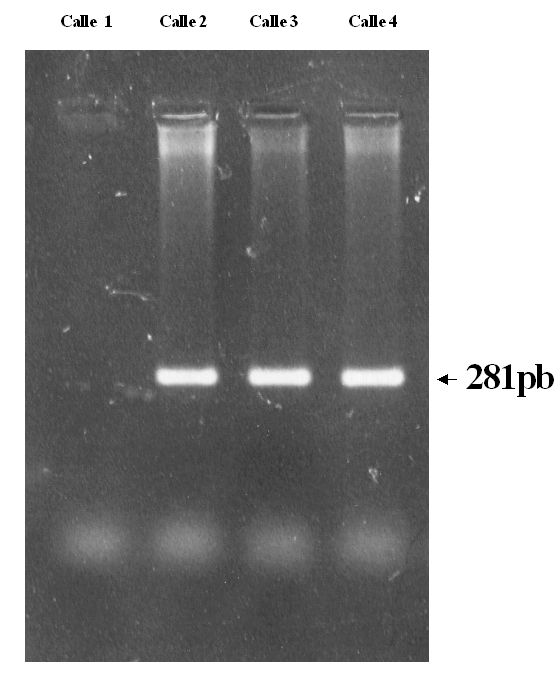
Figura 1: Recorrido electroforético en gel de agarosa al 2% del producto de amplificación por PCR
del fragmento de 281 pb del gen del SRI -1.
Calle 1: control negativo de PCR. Calle 2: rata Wistar. Calle 3: rata eSS. Calle 4: control humano.
DISCUSIÓN
Debido a la estrecha sintenia entre humanos y roedores, se ha sugerido que el estudio de diferentes cepas endocriadas puede ayudar a la identificación de genes candidatos para la diabetes en seres humanos20, entre ellos los que están involucrados en la señalización de la insulina y la insulinoresistencia21-22.
Los resultados verificados permiten concluir que la línea endocriada eSS no presenta la variante Phe-809 en el gen de IRS-1, por lo que puede descartársela como causante de su insulinorresistencia.
Cabe señalar que si bien pacientes portadores de distintas mutaciones del IRS-1 muestran anormalidades metabólicas propias de síndromes de resistencia a la insulina, las mismas suelen ser marcadas8 lo que difiere del síndrome diabético de la línea eSS, caracterizado por ser de moderada intensidad13. Asimismo, los modelos animales con resistencia a la insulina debida a defectos genéticos inducidos en el IRS-1 tampoco son semejantes al síndrome diabético de eSS.
Es pertinente señalar que la diabetes de la rata eSS surgió en forma súbita en la colonia de ratas eIIM de la Cátedra de Biología de la Facultad de Ciencias Médicas de Rosario afectando rápidamente a toda la descendencia13 por lo que se sugirió como factor etiológico un complejo reordenamiento genético producto de su elevada endocría, más que la aparición súbita de una mutación puntual23. Los resultados obtenidos avalan esta hipótesis.
AGRADECIMIENTOS:
Los autores agradecen a los doctores Gustavo Frechtel y Ariel López por sus consejos en la elaboración del trabajo.
REFERENCIAS
- 1. Malecki MT. Genetics of type 2 diabetes mellitus. Diabetes Res Clin Pract 2005; 68 (Suppl1): S10-S21.
2. Reaven GM. Role of insulin resistance in human disease. Banting lecture 1988. Diabetes 1988; 37: 1595-1607.
3. Kadowaki T. Insights into insulin resistance and type 2 diabetes from knockout mouse models. J Clin Invest 2000; 106: 459-465.
4. Rung J, Cauchi S, Albrechtsen A, Shen L, Rocheleau G, Cavalcanti-Proença C, Bacot F, Balkau B, Belisle A, Borch-Johnsen K, Charpentier G, Dina C, Durand E, Elliott P, Hadjadj S, Järvelin MR, Laitinen J, Lauritzen T, Marre M, Mazur A, Meyre D, Montpetit A, Pisinger C, Posner B, Poulsen P, Pouta A, Prentki M, Ribel-Madsen R, Ruokonen A, Sandbaek A, Serre D, Tichet J, Vaxillaire M, Wojtaszewski JF, Vaag A, Hansen T, Polychronakos C, Pedersen O, Froguel P, Sladek R R. Genetic variant near IRS1 is associated with type 2 diabetes, insulin resistance and hyperinsulinemia. Nat Genet 2009; 41: 1110-1115.
5. Sesti G, Federici M, Hribal ML, Lauro D, Sbraccia P, Lauro R. Defects of the insulin receptor substrate (IRS) system in human metabolic disorders. FASEB J 2001; 15: 2099-2111.
6. Chou CK, Dull TJ, Russel DS, Gherzi R, Lewohl D, Ullrich A, Rosen OM. Human insulin receptors mutated at the ATP-binding site lack protein tyrosine kinase activity and fall to mediate postreceptor effects of insulin. J Biol Chem 1987; 262: 1842-1847.
7. Sun XJ, Rothenberg P, Kahn CR, Backer JM, Araki E, Wilden PA, Cahill DA, Goldstein BJ, White MF. Structure of the insulin receptor substrate IRS-1 defines a unique signal transduction protein. Nature 1991; 352: 73-77.
8. Berger D, Barroso I, Soos M, Yeo G, Schafer AJ, O'Rahilly S, Whitehead JP. Genetic variants of insulin receptor substrate-1 (IRS-1) in syndromes of severe insulin resistance. Functional analysis of Ala513Pro and Gly1158Glu IRS-1. Diabetic Medicine 2002; 19: 804-809.
9. Zick Y. Role of Ser/Thr kinases in the uncoupling of insulin signaling. Int J Obes Relat Metab Disord 2003; 27 (Suppl 3): S56-60.
10. Celi FS, Negri C, Tanner K, Raben N, De Pablo F, Rovira A, Pallardo LF, Martin-Vaquero P, Stern MP, Mitchell BD, Shuldiner AR. Molecular scanning for mutations in the insulin receptor substrate-1 (IRS-1) gene in Mexican Americans with Type 2 diabetes mellitus Diabetes Metab Res Rev 2000; 16:370-377.
11. Tamemoto H, Kadowaki T, Tobe K, Yagi T, Sakura H, Hayakawa T, Terauchi Y, Ueki K, Yaburagi Y, Satoh S, Sekihara H, Yoshioka S, Horikoshi H, Furuta Y, Ikawa Y, Kasuga M, Yazaki Y, Aizawa S. Insulin resistance and growth retardation in mice made with targeted disruption of the IRS-1 gene. Nature 1994; 372: 182-186.
12. Kido Y, Burks DJ, Withers D, Bruning JC, Kahn CR, White MF, Accili D. Tissue-specific insulin resistance in mice with mutations in the insulin receptor, IRS-1, and IRS-2. J Clin Invest 2000; 105: 199-205.
13. Martínez SM, Tarrés MC, Montenegro SM, Milo R, Picena JC, Figueroa N, Rabasa SL. Spontaneous diabetes in eSS rats. Acta diabetol lat 1988; 25: 303-313.
14. de Gómez Dumm INT, Montenegro S, Tarrés MC, Martínez SM, Igal RA. Early lipid alterations in spontaneously diabetic rats. Acta Physiol Pharmacol Ther Latinoam 1998; 48: 228-234.
15. Picena JC, Montenegro SM, Tarrés MC, Martínez SM. Modificaciones dinámicas en los islotes de Langerhans de dos líneas de ratas espontáneamente diabéticas: eSS y eSMT. Medicina (Buenos Aires) 2007; 67: 331-340.
16. Tarrés MC, Martínez SM, Montenegro SM, Picena JC, Llorens A, Naves A. The eSS rat: A new model of non-insulin-dependent human diabetes. Am J Pathol 1992; 761-763.
17. Sambrook J, Russell DW Molecular Cloning: A Laboratory Manual. Manniatis T, Sambrook, eds. Cold Spring Harbor Laboratory Press. Third Edition. Cold Spring Harbor, NY, 2001, vol. 1, p. 1-83.
18. Davis LG, Dibner M, Battery JF. Basic methods in molecular biology, Elsevier Science Publishing, New York, 1986, p. 42-50.
19. Maniatis T, Fritsch EF, Sambrook J. Molecular cloning: A laboratory manual. Cold Spring Harbor Laboratory Press, Cold Spring Harbor, NY, 1982, p. 191-195.
20. Ktorza A, Bernard C, Parent V, Penicaud L, Froguel P, Lathrop M, Gauguier D. Are animal models of diabetes relevant to the study of the genetics of non-insulin-dependent diabetes in humans? Diabetes Metab 1997; 23 (Suppl 2): 38-46.
21. Baudry A, Loic L, Jackerott M, Joshi R. Genetic manipulation of insulin signaling, action and secretion in mice. EMBO reports 3: 323-328, 2002.
22. Nandi A, Kitamura Y, Kahn CR, Accili D. Mouse models of insulin resistance. Physiol Rev 2004; 84, 623-647.
23. Calderari S, Martínez SM, Tarrés MC, Picena JC, Rabasa SL: Modelos hereditarios de diabetes y obesidad en líneas endocriadas vinculadas por ascendencia. Mendeliana 1995; 11: 47-55.
CORRESPONDENCIA:
Stella Maris Martínez.
Cátedra de Biología, Facultad de Ciencias Médicas
Universidad Nacional de Rosario.
Santa Fe 3100.
Rosario (2000), Argentina
E-mail: smartinez948 @ yahoo.com.mx
